
Книгу, состоящую из 53 426 слов, 11 картинок и одной Java-программы, ученым из Гарварда удалось записать в массив ДНК.
Общий объем данных, записанных с помощью ДНК, составил 5,27 мегабит
Ученым из Гарварда удалось записать в массив ДНК целую книгу, состоящую из 53 426 слов, 11 картинок и одной Java-программы. Стоимость расшифровки ДНК ежегодно уменьшается примерно в 5—12 раз, и это намного быстрее, чем падение стоимости цифрового электронно-оптического мегабайта. Так что технологию ДНК-памяти ждет большое будущее.
Молекулы ДНК, содержащие детальные инструкции, необходимые для роста, размножения и поддержания жизни организмов, относятся к одним из самых миниатюрных, емких и стабильных носителей информации, оставляющих далеко позади все искусственно созданные на настоящий момент устройства памяти. Идея использовать ДНК для записи, хранения и считывания данных обсуждается и исследуется уже давно, притом не только в научно-фантастических романах, но и в научной периодике, где регулярно появляются сообщения об испытании очередной экспериментальной технологии ДНК-памяти.
Технологический потенциал этой молекулы действительно очень впечатляет.
В первую очередь это относится к плотности упаковки данных: теоретически, ДНК может кодировать два бита информации на один нуклеотид (повторяющиеся блоки, из которых состоит молекула), что в результате дает огромное число — 455 эксабайт (1 эксабайт = 1018 байт) данных на один грамм одноцепочечной ДНК. Такого грамма вполне хватило бы, чтобы записать весь среднегодовой объем глобального интернет-трафика за 2012 год.
Вторым преимуществом ДНК-памяти является ее стабильность: в отличие от цифровых магнитных и оптических носителей, реальное время жизни которых прогнозировать довольно сложно из-за молодости и несовершенства технологии, информация, записанная в ДНК посредством химических связей, может храниться десятки тысяч лет, что уже позволяет считывать генотипы некоторых вымерших в незапамятные времена животных и растений, а с дальнейшим развитием технологии — и возвращать их к жизни.
Наконец, универсальный энзимный механизм записи и считывания информации, отшлифованный за миллиарды лет эволюции живого вещества, позволяет рассматривать ДНК-память в качестве будущего потенциального стандарта хранения и считывания данных.
Ведь почти идеальное устройство памяти уже создано самой природой, роль же технологии сводится к тому, как именно его использовать.
Первое искусственное устройство ДНК-памяти было продемонстрировано еще в 1988 году, когда с помощью этой молекулы удалось закодировать 7920 битов данных. Но по сравнению с этим достижением технология, предложенная исследовательской группой, работающей под руководством Джорджа Чёрча с факультета генетики Гарвардской медицинской школы, выглядит уже настоящим прорывом: используя совершенно новый подход в кодировании информации на ДНК, а также ДНК-синтезаторы и секвенаторы новейшего поколения, генетикам удалось «затолкать» в массив одноцепочечных ДНК целую книгу в формате HTML объемом в 53 426 слов, а кроме того, 11 изображений в формате JPG и одну программу, написанную на языке Java.
Общий объем данных, записанных с помощью ДНК, составил 5,27 мегабит.
Для кодирования 5,27-мегабитного массива группа Чёрча использовала искусственно синтезированные последовательности нуклеотидов, так называемые олигонуклеотиды (форма нуклеиновой кислоты, содержащая относительно небольшое, до нескольких десятков, число нуклеотидов). Каждый олигонуклеотид включал в себя 96-битный блок данных (96 нуклеотидов), 19-битный адрес, по которому определялось место блока в общем массиве (19 нуклеотидов), и однообразные 22-битные технические последовательности оснований, необходимые для последующей амплификации и финальной расшифровки молекулярной записи с помощью ДНК-секвенатора (амплификация — создание дополнительных копий цепочек нуклеотидов, необходимых для последующих манипуляций с информацией, содержащейся в ДНК, — например, сравнительной коррекции и сведения к минимуму ошибок при ее расшифровке).
В общей сложности для записи 5,27-мегабитного массива было использовано 54 898 159 нуклеотидов, организованных в 115-битные олигонуклеотидные блоки.
Последние были синтезированы обычным образом с помощью специальных полностью автоматизированных установок, позволяющих поэтапно выстраивать олигонуклеотидные цепочки с определенной последовательностью оснований, при этом аденин и цитозин принимались за условный ноль, а гуанин и тимин — за условную единицу.
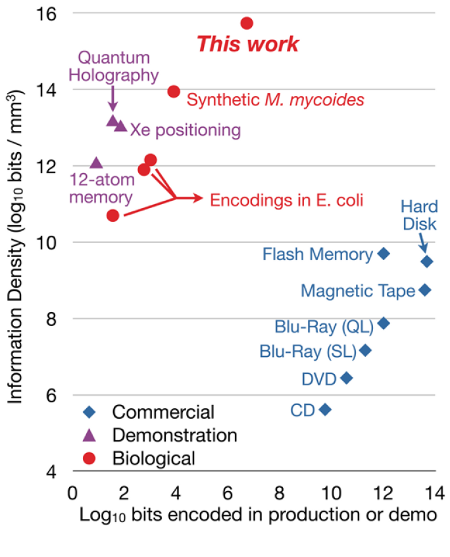
Емкостные характеристики устройств памяти — экспериментальных и коммерческих: вертикальная координата — плотность записи , горизонтальная — емкость носителя (шкалы логарифмические). Красная точка сверху — ДНК-память, разработанная группой Чёрча. // G.Church et al.
Таким образом, разбив массив на относительно небольшие 115-битные блоки олигонуклеотидов, удалось избежать необходимости синтезировать длинные ДНК-последовательности для кодирования больших объемов информации, тем более что современные установки способны выстраивать точные цепочки из, как правило, не более 200 нуклеотидов.
Все это упростило последующую расшифровку и коррекцию ошибок, также осуществленных по обычной схеме с помощью автоматизированной полимеразно-цепной реакции и параллельных ДНК-секвенаторов новейшего поколения: ДНК-цепочки многократно клонировали, далее, скорректировав ошибки с помощью «зеркальных» цепочек, прочитали, а получившиеся коды соединили в массив данных в соответствии с адресными метками.
Теоретическая плотность ДНК-записи, достигаемая с помощью подобной технологии, составляет 5,5 петабит (1 петабит = 1015) на один кубический миллиметр.
Это беспрецедентно высокий показатель для устройств памяти, притом не только коммерческих, но также экспериментальных, например квантово-голографических.
Тем не менее практическое использование этого метода сильно ограничено его громоздкостью, продолжительностью цикла запись/чтение и, конечно, стоимостью.
Впрочем, стоимость расшифровки ДНК ежегодно падает примерно в 5—12 раз — намного быстрей, чем стоимость цифрового электронно-оптического мегабайта, так что у технологии ДНК-памяти, безусловно, есть большое будущее, на которое намекает и название закодированной в ДНК книги — «Регенезис: Как синтетическая биология заново откроет природу и нас самих» (´Regenesis: How Synthetic Biology Will Reinvent Nature and Ourselves´, Basic Books, New York), одним из авторов которой является Джордж Чёрч.
Мечту записали в ДНК

В искусственную ДНК записали пять файлов общим объемом 5,2 мегабита
ДНК поможет людям сохранять огромные массивы информации
В искусственную ДНК записали пять файлов общим объемом 5,2 мегабита, в том числе звуковой файл с речью Мартина Лютера Кинга и научную статью нобелевских лауреатов, расшифровавших структуру этой молекулы.
Объединенная исследовательская группа из Европейского института биоинформатики (EBI), расположенного в Великобритании, и Европейской лаборатории молекулярной биологии (EMBL), расположенной в Германии, совместно с компанией Agilent Technologies (США) разработала технологию, позволяющую использовать искусственные ДНК в качестве долговременного, надежного и энергонезависимого носителя информации. Статья с описанием технологии опубликована сегодня в Nature.
Используя в качестве устройства памяти короткие одноцепочечные ДНК, так называемые олигонуклеотиды (олигонуклеотид — короткая форма нуклеиновой кислоты, содержащая относительно небольшое, до нескольких десятков, число нуклеотидов), исследователи записали на массив таких ДНК пять различных файлов, содержащих полное собрание сонетов Шекспира (текст в формате ASCII), статью первооткрывателей структуры ДНК Джеймса Уотсона и Френсиса Крика «Молекулярная структура нуклеиновых кислот» в формате PDF, цветное фото здания ЕBI в формате JPEG, 26-секундный MP3-файл с фрагментом речи Мартина Лютера Кинга «У меня есть мечта», а также файл с алгоритмом Хаффмана, использованным для конвертации бинарных файлов в вид, удобный для представления данных через последовательность азотистых оснований ДНК.
Общий объем полезных данных, записанных и считанных с ДНК, составил примерно 5,2 мегабита.

Доктор Ник Голдман из EMBL-EBI держит в руках пробирку со всеми сонетами Шекспира, классической научной статьей, звуковым файлом и фотографией своего института, записанными на ДНК. // Nature
Для записи этого объема было использовано 153 335 синтезированных коротких цепочек ДНК по 117 нуклеотидов (117 битов) каждая. Данные кодировались в четырех блоках по 25 нуклеотидов. В оставшихся 17 нуклеотидах (17 бит) кодировались адресные метки, необходимые для сборки данных в исходный файловый массив.
Кодирование происходило в три этапа. Двоичный код, в котором были представлены данные, сначала конвертировался на компьютере в троичный посредством алгоритма Хаффмана, с помощью которого восьмибитные блоки данных (байты) представлялись в виде последовательности из пяти троичных чисел, или тритов (0,1,2). Далее блочная последовательность тритов конвертировалась в код из трех нуклеотидов.
Троичная кодировка позволяла не только сжать данные, но и уменьшить вероятность ошибок при последующем считывании ДНК и реконструкции двоичного массива.
Как известно, ДНК представляет собой полимерную молекулу, в состав которой входят четыре нуклеотида (аденин, гуанин, тимин и цитозин — А, Г, Т, Ц). Для конвертации троичного кода достаточно трех, поэтому в каждом последующем троичном блоке основания можно было комбинировать по-разному, ведь один из четырех нуклеотидов в них мог отсутствовать. Последнее гарантировало, что при синтезе ДНК два и более одинаковых нуклеотида не пришлось бы стыковать в одну полимерную цепочку (так называемый гомополимер), что снижает вероятность ошибок при последующей реконструкции данных.
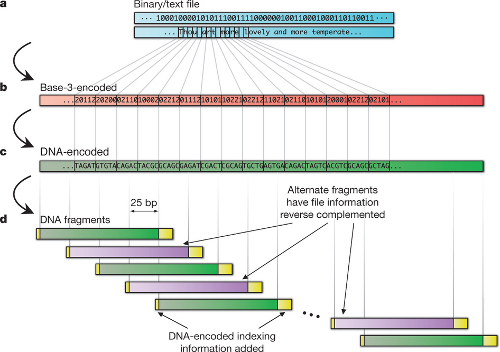
Схема конвертации данных (сонета Шекспира) в ДНК-массив: а) двоичный код b) троичный код c) ДНК-код d) дублированные фрагменты ДНК с шаговым смещением 25 бит (желтым отмечены участки ДНК с адресными метками). // Nature
Полученные таким образом 153335 ДНК-кода были отосланы в США в Agilent Technologies, где они были синтезированы на специальном оборудовании, при этом каждая из 117-битных олигонуклеотидных молекул была размножена в 12 млн копий.
Замороженный и высушенный в вакууме массив синтезированных ДНК, представляющий собой крошечную щепотку органики в герметично запаянной пробирке, был отослан обычной срочной почтой обратно в Англию и далее — в Германию, в одну из лабораторий EMBL, где ДНК были обратно расшифрованы с почти стопроцентной точностью, позволившей, в свою очередь, успешно реконструировать пять первоначальных файлов (число и содержание которых сотрудники лаборатории не знали).
Рассматривать ДНК-память в качестве будущего потенциального стандарта хранения и считывания данных позволяют впечатляющие преимущества, которые имеет эта технология перед электронно-оптическими запоминающими устройствами, которые используются сейчас. Это огромная плотность записи (теоретически, то есть в предельном «идеальном» случае в одном грамме одноцепочечной ДНК можно записать до 455 эксабайт данных, кодируя два бита на один нуклеотид), энергонезависимость, а также долговечность: ДНК со временем хоть и деградирует, но в природной среде может сохранять информацию десятки тысяч лет, а при искусственной консервации и дольше.
Запоминать информацию посредством ДНК успешно пробуют еще с конца 80-х, однако настоящий прорыв в этом направлении произошел только сейчас, со стремительным удешевлением и, главное, увеличением точности технологий по быстрому синтезу и расшифровке ДНК-молекул.
Заметим, что команда EBI-EMBL, описавшая технологию своей ДНК-памяти в Nature, не является здесь первопроходцем.
Относительно недавно группа Джорджа Чёрча, давно экспериментирующая с ДНК-памятью и работающая в Гарварде, сообщила в конкурирующем Science, что ей удалось записать и считать с синтезированного массива коротких одноцепочечных ДНК несколько файлов (книгу, изображения и JAVA-код), притом точно такого же общего объема — 5,2 мегабита, о чем еще полгода назад подробно писала «Газета.Ru».
Сравнение использованных технологий показывает, что обе группы использовали практически идентичные методы записи и считывания информации с ДНК.
Массив данных сначала разбивался на блоки размером чуть больше ста бит, затем перекодировался в буквенную последовательность нуклеотидов, на основе которой синтезировались короткие, чуть больше 100 оснований, ДНК-цепочки. Считывание информации с массива осуществлялось с помощью автоматизированной полимеразно-цепной реакции и параллельных ДНК-секвенаторов новейшего поколения: ДНК-цепочки многократно клонировали, далее, одновременно корректируя ошибки, прочитывали, а получившиеся коды соединяли в массивы данных в соответствии с адресными метками, расположенными на концах цепочек.
Единственное существенное отличие заключается в схеме кодирования двоичного потока в последовательность нуклеотидов: если группа Чёрча использовала простую схема конвертации, приняв пару разных оснований (например, АГ и ТЦ) за условные «ноль» и «единицу», то команда EBI-EMBL использовала более сложный алгоритм, конвертировав битовый поток в тритовый (троичный) посредством алгоритма Хаффмана. Последнее позволило сжать данные, затолкав больше информации в 5,2 мегабит, и снизить вероятность ошибок, исключив из ДНК-массива гомополимерные цепочки. Еще одним трюком, повысившим устойчивость к ошибкам, было четырехкратное дублирование 117-битных цепочек с регулярным смещением кода на 25 бит, притом каждый второй дубль кодировался в обратной последовательности. При такой схеме вероятность возникновения одинаковых ошибок сразу в нескольких цепочках становится ничтожно малой.
Именно устойчивость к ошибкам авторы статьи в Narture назвали главным преимуществом своей технологии, отвечая на специально организованном пресс-брифинге на вопрос, чем же их ДНК-память отличается от ДНК-памяти, разработанной в Гарварде.
С этим, впрочем, можно и поспорить: во-первых, группа Чёрча также заложила в свою ДНК-память алгоритм коррекции ошибок, при котором сравнивались коды размноженных «зеркальных» ДНК-цепочек. Во-вторых, сами авторы статьи в Nature признают «избыточность» своей схемы, так как точность современных устройств, синтезирующих и считывающих короткие, до 200 оснований, цепочки ДНК, очень высокая, а среднее число ошибок редко превышает одну на 500 оснований.

Фотография EBI, записанная и считанная с помощью ДНК. // Nature
Как бы то ни было, несмотря на идентичность проведенных опытов по эксплуатации искусственной ДНК в качестве носителя данных, а также забавные издержки конкуренции двух главных научных журналов, державших в секрете друг от друга почти одинаковые по содержанию статьи с описанием интересной и перспективной технологии, которые поступили к ним почти в одно и то же время — в начале лета 2012 года (Science, как видим, отреагировал более оперативно, и планируемой маленькой сенсации у Nature все-таки не вышло), дебют ДНК-памяти можно считать успешным. Потенциальной же областью ее применения может стать долгосрочное архивирование относительно нечасто запрашиваемой информации: оценив темпы, с которой дешевеет технология ДНК-синтеза и дешифровки, группа EBI-EMBL прогнозирует, что конкурировать с технологиями хранения данных на магнитных лентах, до сих пор весьма востребованными, ДНК-память сможет уже в ближайшие 50 лет.
http://www.gazeta.ru/science/2012/08/17_a_4729097.shtml
http://www.gazeta.ru/science/2013/01/24_a_4938865.shtml
Общий объем данных, записанных с помощью ДНК, составил 5,27 мегабит
Ученым из Гарварда удалось записать в массив ДНК целую книгу, состоящую из 53 426 слов, 11 картинок и одной Java-программы. Стоимость расшифровки ДНК ежегодно уменьшается примерно в 5—12 раз, и это намного быстрее, чем падение стоимости цифрового электронно-оптического мегабайта. Так что технологию ДНК-памяти ждет большое будущее.
Молекулы ДНК, содержащие детальные инструкции, необходимые для роста, размножения и поддержания жизни организмов, относятся к одним из самых миниатюрных, емких и стабильных носителей информации, оставляющих далеко позади все искусственно созданные на настоящий момент устройства памяти. Идея использовать ДНК для записи, хранения и считывания данных обсуждается и исследуется уже давно, притом не только в научно-фантастических романах, но и в научной периодике, где регулярно появляются сообщения об испытании очередной экспериментальной технологии ДНК-памяти.
Технологический потенциал этой молекулы действительно очень впечатляет.
В первую очередь это относится к плотности упаковки данных: теоретически, ДНК может кодировать два бита информации на один нуклеотид (повторяющиеся блоки, из которых состоит молекула), что в результате дает огромное число — 455 эксабайт (1 эксабайт = 1018 байт) данных на один грамм одноцепочечной ДНК. Такого грамма вполне хватило бы, чтобы записать весь среднегодовой объем глобального интернет-трафика за 2012 год.
Вторым преимуществом ДНК-памяти является ее стабильность: в отличие от цифровых магнитных и оптических носителей, реальное время жизни которых прогнозировать довольно сложно из-за молодости и несовершенства технологии, информация, записанная в ДНК посредством химических связей, может храниться десятки тысяч лет, что уже позволяет считывать генотипы некоторых вымерших в незапамятные времена животных и растений, а с дальнейшим развитием технологии — и возвращать их к жизни.
Наконец, универсальный энзимный механизм записи и считывания информации, отшлифованный за миллиарды лет эволюции живого вещества, позволяет рассматривать ДНК-память в качестве будущего потенциального стандарта хранения и считывания данных.
Ведь почти идеальное устройство памяти уже создано самой природой, роль же технологии сводится к тому, как именно его использовать.
Первое искусственное устройство ДНК-памяти было продемонстрировано еще в 1988 году, когда с помощью этой молекулы удалось закодировать 7920 битов данных. Но по сравнению с этим достижением технология, предложенная исследовательской группой, работающей под руководством Джорджа Чёрча с факультета генетики Гарвардской медицинской школы, выглядит уже настоящим прорывом: используя совершенно новый подход в кодировании информации на ДНК, а также ДНК-синтезаторы и секвенаторы новейшего поколения, генетикам удалось «затолкать» в массив одноцепочечных ДНК целую книгу в формате HTML объемом в 53 426 слов, а кроме того, 11 изображений в формате JPG и одну программу, написанную на языке Java.
Общий объем данных, записанных с помощью ДНК, составил 5,27 мегабит.
Для кодирования 5,27-мегабитного массива группа Чёрча использовала искусственно синтезированные последовательности нуклеотидов, так называемые олигонуклеотиды (форма нуклеиновой кислоты, содержащая относительно небольшое, до нескольких десятков, число нуклеотидов). Каждый олигонуклеотид включал в себя 96-битный блок данных (96 нуклеотидов), 19-битный адрес, по которому определялось место блока в общем массиве (19 нуклеотидов), и однообразные 22-битные технические последовательности оснований, необходимые для последующей амплификации и финальной расшифровки молекулярной записи с помощью ДНК-секвенатора (амплификация — создание дополнительных копий цепочек нуклеотидов, необходимых для последующих манипуляций с информацией, содержащейся в ДНК, — например, сравнительной коррекции и сведения к минимуму ошибок при ее расшифровке).
В общей сложности для записи 5,27-мегабитного массива было использовано 54 898 159 нуклеотидов, организованных в 115-битные олигонуклеотидные блоки.
Последние были синтезированы обычным образом с помощью специальных полностью автоматизированных установок, позволяющих поэтапно выстраивать олигонуклеотидные цепочки с определенной последовательностью оснований, при этом аденин и цитозин принимались за условный ноль, а гуанин и тимин — за условную единицу.

Емкостные характеристики устройств памяти — экспериментальных и коммерческих: вертикальная координата — плотность записи , горизонтальная — емкость носителя (шкалы логарифмические). Красная точка сверху — ДНК-память, разработанная группой Чёрча. // G.Church et al.
Таким образом, разбив массив на относительно небольшие 115-битные блоки олигонуклеотидов, удалось избежать необходимости синтезировать длинные ДНК-последовательности для кодирования больших объемов информации, тем более что современные установки способны выстраивать точные цепочки из, как правило, не более 200 нуклеотидов.
Все это упростило последующую расшифровку и коррекцию ошибок, также осуществленных по обычной схеме с помощью автоматизированной полимеразно-цепной реакции и параллельных ДНК-секвенаторов новейшего поколения: ДНК-цепочки многократно клонировали, далее, скорректировав ошибки с помощью «зеркальных» цепочек, прочитали, а получившиеся коды соединили в массив данных в соответствии с адресными метками.
Теоретическая плотность ДНК-записи, достигаемая с помощью подобной технологии, составляет 5,5 петабит (1 петабит = 1015) на один кубический миллиметр.
Это беспрецедентно высокий показатель для устройств памяти, притом не только коммерческих, но также экспериментальных, например квантово-голографических.
Тем не менее практическое использование этого метода сильно ограничено его громоздкостью, продолжительностью цикла запись/чтение и, конечно, стоимостью.
Впрочем, стоимость расшифровки ДНК ежегодно падает примерно в 5—12 раз — намного быстрей, чем стоимость цифрового электронно-оптического мегабайта, так что у технологии ДНК-памяти, безусловно, есть большое будущее, на которое намекает и название закодированной в ДНК книги — «Регенезис: Как синтетическая биология заново откроет природу и нас самих» (´Regenesis: How Synthetic Biology Will Reinvent Nature and Ourselves´, Basic Books, New York), одним из авторов которой является Джордж Чёрч.
Мечту записали в ДНК

В искусственную ДНК записали пять файлов общим объемом 5,2 мегабита
ДНК поможет людям сохранять огромные массивы информации
В искусственную ДНК записали пять файлов общим объемом 5,2 мегабита, в том числе звуковой файл с речью Мартина Лютера Кинга и научную статью нобелевских лауреатов, расшифровавших структуру этой молекулы.
Объединенная исследовательская группа из Европейского института биоинформатики (EBI), расположенного в Великобритании, и Европейской лаборатории молекулярной биологии (EMBL), расположенной в Германии, совместно с компанией Agilent Technologies (США) разработала технологию, позволяющую использовать искусственные ДНК в качестве долговременного, надежного и энергонезависимого носителя информации. Статья с описанием технологии опубликована сегодня в Nature.
Используя в качестве устройства памяти короткие одноцепочечные ДНК, так называемые олигонуклеотиды (олигонуклеотид — короткая форма нуклеиновой кислоты, содержащая относительно небольшое, до нескольких десятков, число нуклеотидов), исследователи записали на массив таких ДНК пять различных файлов, содержащих полное собрание сонетов Шекспира (текст в формате ASCII), статью первооткрывателей структуры ДНК Джеймса Уотсона и Френсиса Крика «Молекулярная структура нуклеиновых кислот» в формате PDF, цветное фото здания ЕBI в формате JPEG, 26-секундный MP3-файл с фрагментом речи Мартина Лютера Кинга «У меня есть мечта», а также файл с алгоритмом Хаффмана, использованным для конвертации бинарных файлов в вид, удобный для представления данных через последовательность азотистых оснований ДНК.
Общий объем полезных данных, записанных и считанных с ДНК, составил примерно 5,2 мегабита.

Доктор Ник Голдман из EMBL-EBI держит в руках пробирку со всеми сонетами Шекспира, классической научной статьей, звуковым файлом и фотографией своего института, записанными на ДНК. // Nature
Для записи этого объема было использовано 153 335 синтезированных коротких цепочек ДНК по 117 нуклеотидов (117 битов) каждая. Данные кодировались в четырех блоках по 25 нуклеотидов. В оставшихся 17 нуклеотидах (17 бит) кодировались адресные метки, необходимые для сборки данных в исходный файловый массив.
Кодирование происходило в три этапа. Двоичный код, в котором были представлены данные, сначала конвертировался на компьютере в троичный посредством алгоритма Хаффмана, с помощью которого восьмибитные блоки данных (байты) представлялись в виде последовательности из пяти троичных чисел, или тритов (0,1,2). Далее блочная последовательность тритов конвертировалась в код из трех нуклеотидов.
Троичная кодировка позволяла не только сжать данные, но и уменьшить вероятность ошибок при последующем считывании ДНК и реконструкции двоичного массива.
Как известно, ДНК представляет собой полимерную молекулу, в состав которой входят четыре нуклеотида (аденин, гуанин, тимин и цитозин — А, Г, Т, Ц). Для конвертации троичного кода достаточно трех, поэтому в каждом последующем троичном блоке основания можно было комбинировать по-разному, ведь один из четырех нуклеотидов в них мог отсутствовать. Последнее гарантировало, что при синтезе ДНК два и более одинаковых нуклеотида не пришлось бы стыковать в одну полимерную цепочку (так называемый гомополимер), что снижает вероятность ошибок при последующей реконструкции данных.

Схема конвертации данных (сонета Шекспира) в ДНК-массив: а) двоичный код b) троичный код c) ДНК-код d) дублированные фрагменты ДНК с шаговым смещением 25 бит (желтым отмечены участки ДНК с адресными метками). // Nature
Полученные таким образом 153335 ДНК-кода были отосланы в США в Agilent Technologies, где они были синтезированы на специальном оборудовании, при этом каждая из 117-битных олигонуклеотидных молекул была размножена в 12 млн копий.
Замороженный и высушенный в вакууме массив синтезированных ДНК, представляющий собой крошечную щепотку органики в герметично запаянной пробирке, был отослан обычной срочной почтой обратно в Англию и далее — в Германию, в одну из лабораторий EMBL, где ДНК были обратно расшифрованы с почти стопроцентной точностью, позволившей, в свою очередь, успешно реконструировать пять первоначальных файлов (число и содержание которых сотрудники лаборатории не знали).
Рассматривать ДНК-память в качестве будущего потенциального стандарта хранения и считывания данных позволяют впечатляющие преимущества, которые имеет эта технология перед электронно-оптическими запоминающими устройствами, которые используются сейчас. Это огромная плотность записи (теоретически, то есть в предельном «идеальном» случае в одном грамме одноцепочечной ДНК можно записать до 455 эксабайт данных, кодируя два бита на один нуклеотид), энергонезависимость, а также долговечность: ДНК со временем хоть и деградирует, но в природной среде может сохранять информацию десятки тысяч лет, а при искусственной консервации и дольше.
Запоминать информацию посредством ДНК успешно пробуют еще с конца 80-х, однако настоящий прорыв в этом направлении произошел только сейчас, со стремительным удешевлением и, главное, увеличением точности технологий по быстрому синтезу и расшифровке ДНК-молекул.
Заметим, что команда EBI-EMBL, описавшая технологию своей ДНК-памяти в Nature, не является здесь первопроходцем.
Относительно недавно группа Джорджа Чёрча, давно экспериментирующая с ДНК-памятью и работающая в Гарварде, сообщила в конкурирующем Science, что ей удалось записать и считать с синтезированного массива коротких одноцепочечных ДНК несколько файлов (книгу, изображения и JAVA-код), притом точно такого же общего объема — 5,2 мегабита, о чем еще полгода назад подробно писала «Газета.Ru».
Сравнение использованных технологий показывает, что обе группы использовали практически идентичные методы записи и считывания информации с ДНК.
Массив данных сначала разбивался на блоки размером чуть больше ста бит, затем перекодировался в буквенную последовательность нуклеотидов, на основе которой синтезировались короткие, чуть больше 100 оснований, ДНК-цепочки. Считывание информации с массива осуществлялось с помощью автоматизированной полимеразно-цепной реакции и параллельных ДНК-секвенаторов новейшего поколения: ДНК-цепочки многократно клонировали, далее, одновременно корректируя ошибки, прочитывали, а получившиеся коды соединяли в массивы данных в соответствии с адресными метками, расположенными на концах цепочек.
Единственное существенное отличие заключается в схеме кодирования двоичного потока в последовательность нуклеотидов: если группа Чёрча использовала простую схема конвертации, приняв пару разных оснований (например, АГ и ТЦ) за условные «ноль» и «единицу», то команда EBI-EMBL использовала более сложный алгоритм, конвертировав битовый поток в тритовый (троичный) посредством алгоритма Хаффмана. Последнее позволило сжать данные, затолкав больше информации в 5,2 мегабит, и снизить вероятность ошибок, исключив из ДНК-массива гомополимерные цепочки. Еще одним трюком, повысившим устойчивость к ошибкам, было четырехкратное дублирование 117-битных цепочек с регулярным смещением кода на 25 бит, притом каждый второй дубль кодировался в обратной последовательности. При такой схеме вероятность возникновения одинаковых ошибок сразу в нескольких цепочках становится ничтожно малой.
Именно устойчивость к ошибкам авторы статьи в Narture назвали главным преимуществом своей технологии, отвечая на специально организованном пресс-брифинге на вопрос, чем же их ДНК-память отличается от ДНК-памяти, разработанной в Гарварде.
С этим, впрочем, можно и поспорить: во-первых, группа Чёрча также заложила в свою ДНК-память алгоритм коррекции ошибок, при котором сравнивались коды размноженных «зеркальных» ДНК-цепочек. Во-вторых, сами авторы статьи в Nature признают «избыточность» своей схемы, так как точность современных устройств, синтезирующих и считывающих короткие, до 200 оснований, цепочки ДНК, очень высокая, а среднее число ошибок редко превышает одну на 500 оснований.

Фотография EBI, записанная и считанная с помощью ДНК. // Nature
Как бы то ни было, несмотря на идентичность проведенных опытов по эксплуатации искусственной ДНК в качестве носителя данных, а также забавные издержки конкуренции двух главных научных журналов, державших в секрете друг от друга почти одинаковые по содержанию статьи с описанием интересной и перспективной технологии, которые поступили к ним почти в одно и то же время — в начале лета 2012 года (Science, как видим, отреагировал более оперативно, и планируемой маленькой сенсации у Nature все-таки не вышло), дебют ДНК-памяти можно считать успешным. Потенциальной же областью ее применения может стать долгосрочное архивирование относительно нечасто запрашиваемой информации: оценив темпы, с которой дешевеет технология ДНК-синтеза и дешифровки, группа EBI-EMBL прогнозирует, что конкурировать с технологиями хранения данных на магнитных лентах, до сих пор весьма востребованными, ДНК-память сможет уже в ближайшие 50 лет.
http://www.gazeta.ru/science/2012/08/17_a_4729097.shtml
http://www.gazeta.ru/science/2013/01/24_a_4938865.shtml












Комментарии (0)